化学品对经济发展和人类生活质量改善至关重要。近年来,我国化学品市场需求不断增长,化学品销售额预计将于2030年占全球近一半[1]。然而,化学品的潜在危害性会对生态和人体健康构成严重威胁[1-3]。有必要对化学品进行合理管控,以保障公众健康和环境安全,实现社会可持续发展。
风险评价是对化学品进行健全管理和污染防治的前提,其关键在于化学品的暴露和危害性评价。化学品暴露是导致风险的前提,可分为外暴露和内暴露。外暴露一般指环境暴露,由化学品的源释放及其环境迁移分配、降解转化行为所决定;内暴露即化学品在生物体内或者特定靶点的暴露,与危害性直接关联;危害性指化学品在特定暴露条件下对生物体或生态系统产生有害效应的能力[4]。传统的化学品风险评价方法依赖于实验测试。然而,由于目前全球市场中注册的化学品及其混合物已超过35万种[5],且化学品的环境行为与毒性效应机制高度复杂,实验测试方法在高效、低成本地获取化学品暴露和危害性参数方面存在局限,难以满足化学品风险评价的需求[6-8]。
计算毒理学技术被视为应对上述挑战的重要工具[9-10]。计算毒理学基于计算化学、系统生物学等原理构建数学或计算机模型,可衔接化学品的源释放量、环境介质浓度、靶点暴露剂量、毒性效应阈值等关键数据,实现不同种类化学品从暴露到导致危害效应连续过程的高通量准确预测。其中,定量构效关系(QSAR)模型可为化学品暴露和效应模拟提供关键参数、填补相关数据空白,是计算毒理学的核心工具[4]。
欧美国家较早认识到计算毒理学方法的应用潜力,积极开展相关研究推动其发展和应用,形成了一系列面向化学品风险预测的计算毒理学软件[11-13]。一些可预测化学品暴露或/和危害性参数的软件已在化学品信息申报、优先测试化学品筛选和绿色化学品研发等活动中得到了广泛应用。本研究选取目前已发展的、可预测化学品环境暴露/危害性质参数的计算毒理学软件作为研究对象,从多个维度对它们进行比较,讨论其开发条件、应用现状和未来的进步空间,以期为我国在化学品管理中发展和应用此类软件或平台提供参考。
1 材料与方法(Materials and methods)
1.1 软件搜集
本研究系统地搜集并选取国内外25款可预测化学品暴露/危害性的计算毒理学软件进行比较研究。通过浏览软件网页介绍和阅读用户指导手册,统计软件的所属国别、授权方式(免费/商用)、发行方式(单机/网络)、发行时间和主要开发者等基本信息。
1.2 预测终点统计
“终点(endpoint)”指在特定实验条件下被测量和建模的物理化学、环境或生物效应指标[14]。本文考察了不同软件对理化性质、环境行为、生态毒性、人体健康毒性和毒代动力学性质参数等5类终点的覆盖情况,并利用Python软件(版本3.9)将结果以热图形式呈现。
1.3 预测方法统计
以软件名称为关键词,在Web of Science检索预测方法相关文献,并利用软件自带的模型介绍和欧盟委员会联合研究中心(JRC)的QSAR模型数据库[15],对软件的预测手段、模型建模方法、分子描述符及应用域表征方法进行统计。
1.4 软件操作
实际操作和试用了12款可免费获取的软件,分别是EPI Suite (版本4.1.1)、OECD QSAR Toolbox (版本4.2.2)、T.E.S.T. (版本4.2.1)、Toxtree (版本3.1.0)、Lazar (版本1.4.2)、OncoLogic (版本8.0)、VEGA (版本1.1.5)、OPERA (版本2.6)、Danish QSAR Database/Models、ProTox Ⅱ、AdmetSAR 2和CPTP。统计了软件具有的功能,并从模型和预测不确定性等方面考察软件的信息完整度。
此外,为了分析上述软件的预测准确度,对软件中提供的模型预测性能信息进行了统计,共统计了正辛醇/水分配系数、鱼类急性毒性、生物富集因子、生物降解性和致突变性模型的内外部验证准确性指标。进一步选择常用的广谱杀菌剂“百菌清”作为预测对象(CAS: 1897-45-6),选择正辛醇/水分配系数(octanol-water partition coefficient, Kow)、黑头软口鲦96 h半数致死浓度(lethal concentration 50%, LC50)(mg·L-1)和鱼类生物富集因子(bioconcentration factor, BCF)(L·kg-1)作为预测终点,比较各软件的预测准确度。
2 结果(Results)
2.1 计算毒理学软件的开发背景
统计的25款软件中,20款由欧洲和美国开发,2款由中国开发,日本、加拿大、经济合作与发展组织(OECD)开发的软件各有1款(表1)。这些软件的开发应用与其服务的国家或地区的化学品管理制度密切相关。EPI Suite、ECOSAR (已被整合于EPI Suite中)和Oncologic等由美国环境保护局(US EPA)开发,辅助其在《有毒物质控制法》(TSCA)下开展化学品风险评价工作。Toxtree、OECD QSAR Toolbox和VEGA等主要服务欧盟的《化学品注册、评估、许可和限制》(REACH)法规,鼓励企业利用计算工具对产品进行风险筛查,或帮助企业填补化学品申报所需信息。Leadscope、CaseUltra、ToxGPS和Derek Nexus等面向美国食品和药物管理局(US FDA),提供满足《人用药品注册技术要求国际协调会(ICH)三方协调导则》要求的预测模型。另外,REACH法规和TSCA对化学品风险评价成本的内部化,催生了面向绿色化工设计的商用软件,如ACD/Percepta、ADMET Predictor和ChemTunes/ToxGPS等。
欧美国家的软件开发参与者跨越了政、商、研各界。首先,欧美国家的政府较早组建了多学科背景的研究团队,并由政府机构如US EPA、JRC等组织收集建模关键数据与模型资源,为监管机构主导开发预测软件提供了充分的条件。发达国家严格的管理法规还催生了一批提供化学品风险评价服务的商业公司,可独立开发预测软件。此外,非盈利性的独立研究团队开发了部分免费软件,如ProTox Ⅱ、AdmetSAR 2等,作为QSAR模型的共享平台。大部分预测软件的开发涉及了政、商、研三方或两两合作,还有在大型国际合作项目下开发的软件如VEGA和OECD QSAR Toolbox,体现了社会各界良好的合作模式以及数据共享机制对计算毒理学软件开发的重要性。统计表明,2款由我国研发的软件:CPTP和AdmetSAR 2,均由高校研究团队独立开发。鉴于计算毒理学软件在化学品风险评价和管理中的先进性和实用性,未来需要弥补我国政府和商业界在此类工具开发中的缺位。
2.2 软件的预测终点
软件的预测终点覆盖度体现了其应用范围(图1)。统计结果表明,上述25款计算毒理学软件的预测终点涵盖了理化性质、环境行为、生态毒性、人体健康毒性和毒代动力学参数等5类。其中,理化性质类参数表征化学品的基本性质,一方面可评估化学品的物理危害,为其储存、运输和使用方式提供参考;另一方面可作为预测化学品环境行为及毒性的重要输入参数;环境行为类参数描述化学品的迁移、转化和归趋,评估化学品的环境持久性或从环境转移到生物体中的潜力;生态毒性终点考察化学品对非人类物种产生危害的能力,评估化学品对生态系统结构和功能造成的影响;人体健康毒性终点包含了化学品对人体不同尺度层级(个体、器官、细胞和生物大分子)的有害效应,评估化学品的人类健康风险;毒代动力学参数包括与毒物在生物体吸收、分布、代谢及排泄过程相关的参数,可预测化学品的生物有效浓度和作用于特定靶点的浓度,辅助体外(in vitro)-体内(in vivo)毒性效应阈值的外推[16]。
统计表明,水溶解度、正辛醇/水分配系数、生物降解性、水生生物积累性、淡水水生生物毒性、体外致突变性和致癌性等是软件中常见的预测终点。相比之下,软件对环境光化学降解性,陆生生物的生物积累性和急性毒性,免疫、神经、生殖毒性,以及大部分毒代动力学参数覆盖较少。不同终点被覆盖次数的差异,可能与法规对该终点的重视程度,对应实验数据的数量和质量,以及对终点相关机理的理解程度有关[13]。
表1 面向化学品风险预测的计算毒理学软件
Table 1 Computational toxicology software for chemical risk prediction

软件Software国家/组织Country/Organization发行方式Release type授权方式License主要开发者Main developers发行时间Release time获取途径AccessEPI Suite美国USA单机Standalone免费FreeUS EPA, Syracuse Inc.1980shttps://www.epa.gov/tsca-screening-tools/down-load-epi-suitetm-estimation-program-interface-v411T.E.S.T.美国USA单机Standalone免费FreeUS EPA2010https://www.epa.gov/chemical-research/toxicity-estimation-software-tool-testPBT Profiler美国USA网络Network免费FreeUS EPA, Syracuse Inc.N/Ahttp://www.pbtprofiler.net/default.aspOncoLogic美国USA单机Standalone免费FreeUS EPA, LogiChem Inc.N/Ahttps://www.epa.gov/tsca-screening-tools/oncolog-ictm-expert-system-evaluate-carcinogenic-potential-chemicalsOECD QSAR Toolbox经合组织OECD单机Standalone免费FreeECHA, OECD, LMC, Lhasa Ltd.2006http://qsartoolbox.orgToxtree欧盟EU单机,网络Standalone, network免费FreeJRC, Ideaconsult Ltd.2007http://toxtree.sourceforge.netToxmatch欧盟EU单机Standalone免费FreeJRC, Ideaconsult Ltd.N/Ahttps://ec.europa.eu/jrc/en/scientific-tool/tox-matchDanish QSAR Database/Models丹麦Denmark网络Network免费FreeTUD, Leadscope Inc.2004http://qsar.food.dtu.dk/VEGA意大利Italy单机Standalone免费FreeIRFMNN/Ahttps://www.vegahub.eu/UL’s Cheminformatics Suite美国USA网络Network商用CommercialUnderwriters Laboratories LLC, JHUN/Ahttps://msc.ul.com/en/products/cheminformatics/Molcode Toolbox爱沙尼亚Estonia单机Standalone商用CommercialMolCode Ltd.N/Ahttps://www.compudrug.com/molcode_toolboxCASE Ultra美国USA单机Standalone商用CommercialMultiCASE Inc.N/Ahttp://multicase.com/Leadscope Model Applier美国USA单机Standalone商用CommercialLeadscope Inc., US FDAN/Ahttps://www.leadscope.com/model_appliers/Derek英国UK单机Standalone商用CommercialLhasa Ltd.1989https://www.lhasalimited.org/products/derek-nex-us.htmACD/Percepta加拿大Canada单机Standalone商用CommercialAdvanced Chemistry Development, Inc.2011https://www.acdlabs.com/products/percepta/index.phpADMET Predictor美国USA单机Standalone商用CommercialSimulations Plus1999https://www.simulations-plus.com/software/admet-predictor/ChemTunes/ToxGPS德国, 美国Germany, USA单机Standalone商用CommercialMolecular Networks GmbH, Altamira LLCN/Ahttps://www.mn-am.com/products/chemtunes-toxgpsProTox Ⅱ德国Germany网络Network免费FreeCU2014http://tox.charite.de/AdmetSAR 2中国China网络Network免费FreeECUST2012http://lmmd.ecust.edu.cn/admetsar2Hazard Expert美国USA单机Standalone商用CommercialCompudrug Ltd.1988https://www.compudrug.com/hazardexpertproBIOVIA美国USA单机Standalone商用CommercialHealth Designs Inc.N/Ahttps://www.3ds.com/products-services/biovia/products/molecular-modeling-simulation/biovia-dis-covery-studio/

软件Software国家/组织Country/Organization发行方式Release type授权方式License主要开发者Main developers发行时间Release time获取途径AccessLazar瑞士Switzerland网络Network免费Freein silico toxicology, BFR2008https://lazar.in-silico.ch/predictOPERA美国USA单机Standalone免费FreeNIEHSN/Ahttp://github.com/NIEHS/OPERAKATE日本Japan单机,网络Standalone, network免费FreeNIES, OU2004https://kate.nies.go.jpCPTP中国China网络Network免费FreeDUT2019http://cptp.dlut.edu.cn/
注:US EPA表示美国环境保护局;ECHA表示欧洲化学品管理局;JRC表示欧盟委员会联合研究中心;US FDA表示美国食品和药物管理局;LMC表示数学化学实验室;TUD表示丹麦技术大学;IRFMN表示马里奥·内格里药理学研究所;JHU表示约翰霍普金斯大学;CU表示查利特大学;ECUST表示华东理工大学;BFR表示德国联邦风险评价研究所;NIEHS表示(美国)国立环境卫生科学研究所;NIES表示(日本)国家环境研究所;OU表示大分大学;DUT表示大连理工大学;N/A表示未知。
Note: US EPA is United States Environmental Protection Agency; ECHA is European Chemicals Agency; JRC is European Commission’s Joint Research Centre; US FDA is United States Food and Drug Administration; LMC is The Laboratory of Mathematical Chemistry; TUD is Technical University of Denmark; IRFMN is Istituto di Ricerche Farmacologiche Mario Negri; JHU is Johns Hopkins University; CU is Charite University; ECUST is East China University of Science and Technology; BFR is The German Federal Institute for Risk Assessment; NIEHS is National Institute of Environmental Health Science; NIES is National Institute for Environmental Studies; OU is Oita University; DUT is Dalian University of Technology; N/A represents unknown.
此外,EPI Suite、PBT Profiler中包含了Ⅲ级多介质环境逸度模型,可结合环境参数、分配参数和降解参数,预测物质在环境各相中的半衰期,有助于评价物质的总体环境持久性[17]。部分软件的人体健康毒性相关终点中涵盖了分子层级的毒性信号通路,如物质与核受体(雌激素、雄激素和甲状腺激素等)的结合能力(Danish QSAR Models, AdmetSAR2, ProTox Ⅱ等)、hERG通道抑制作用(ACD/Percepta, AdmetSAR2, CASE Ultra)等。这些终点对应顶层毒性的分子起始事件(MIEs),可与有害结局路径(AOP)框架结合,从毒性作用机理出发预测化学品潜在疾病风险[18]。
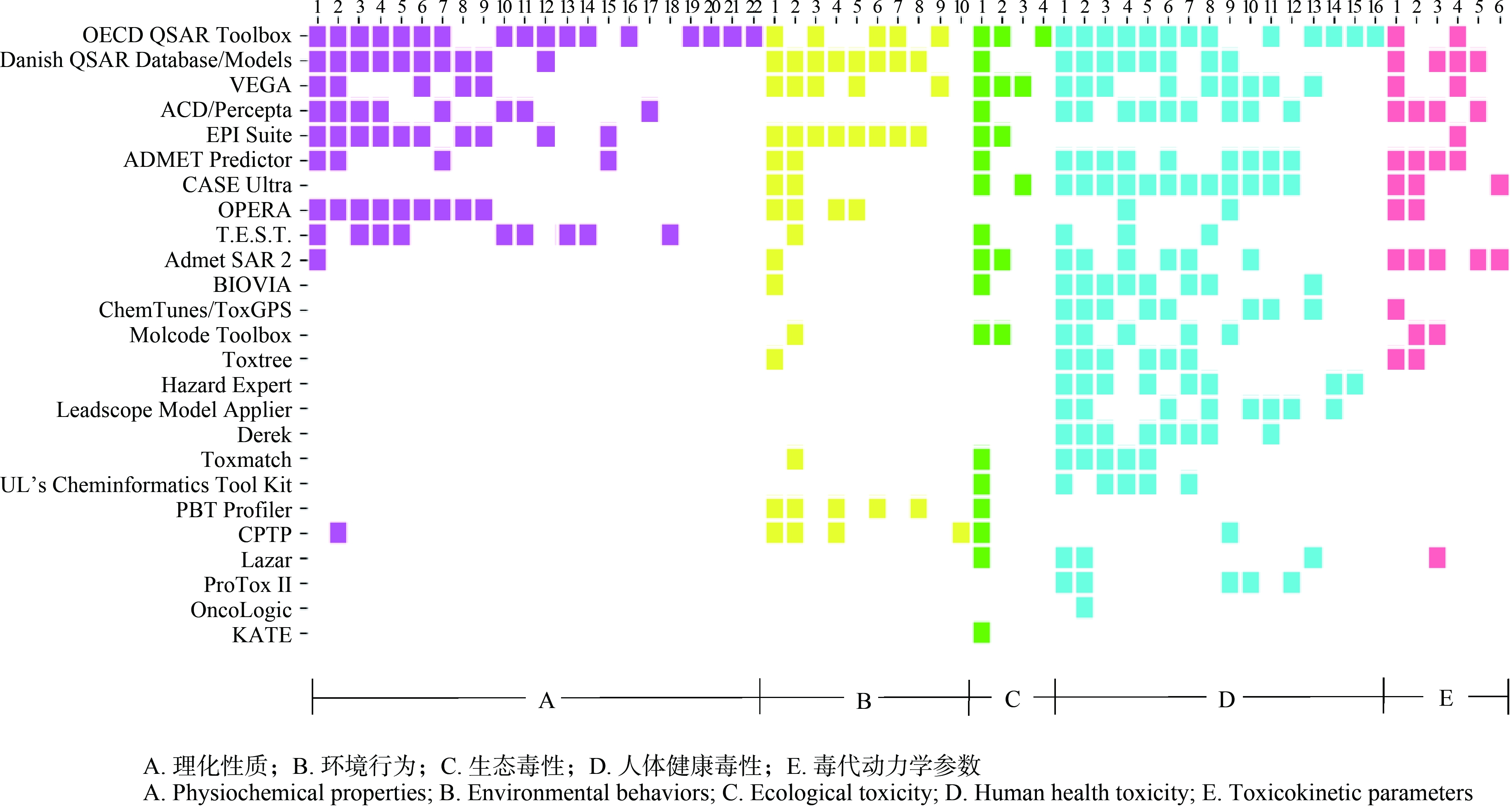
图1 软件涵盖的预测终点
注:A1-水溶解度;A2-正辛醇/水分配系数(Kow);A3-沸点;A4-蒸汽压;A5-熔点;A6-正辛醇/空气分配系数(Koa);A7-解离常数;A8-亨利定律常数;
A9-有机碳吸附系数(Koc);A10-密度;A11-表面张力;A12-颗粒物/空气分配系数(Kp);A13-黏度;A14-闪点;A15-空气/水分配系数(Kaw);
A16-自燃点;A17-极化度;A18-热导率;A19-爆炸性;A20-氧化性;A21-可燃性;A22-脂溶性;B1-生物降解性;B2-生物富集因子(BCF);B3-水解性;
B4-大气氧化性;B5-生物转化速率(鱼);B6-环境相间分配;B7-生物积累因子(BAF);B8-各介质内半衰期;B9-光降解性;B10-生物放大因子(BMF);
C1-水生生物物毒性;C2-陆生生物毒性;C3-微生物抑制作用;C4-沉积物生物毒性;D1-致突变性;D2-致癌性;D3-致敏性;D4-急性毒性;D5-皮肤刺激
性/腐蚀性;D6-基因毒性;D7-眼损伤/刺激性;D8-发育毒性;D9-毒性信号通路;D10-肝毒性;D11-生殖毒性;D12-心脏毒性;D13-重复剂量毒性;
D14-神经毒性;D15-免疫毒性;D16-光致毒性;E1-代谢;E2-DNA/蛋白质结合能力;E3-血脑屏障穿透性;E4-皮肤渗透性;E5-胃肠道吸收;E6-排泄。
Fig.1 Prediction endpoints covered by the software
Note: A1-Water solubility; A2-Octanol-water partition coefficient (Kow); A3-Boiling point; A4-Vapor pressure; A5-Melting point; A6-Octanol-air
partition coefficient (Koa); A7-Dissociation constant; A8-Henry’s Law constant; A9-Organic carbon-sorption partition coefficient (Koc);
A10-Density; A11-Surface tension; A12-Particle-gas partition (Kp); A13-Viscosity; A14-Flashpoint; A15-Air-water partition coefficient (Kaw);
A16-Self-ignition point; A17-Polarizability; A18-Thermal conductivity; A19-Explosive properties; A20-Oxidizing properties; A21-Flammability;
A22-Fat solubility; B1-Biodegradation; B2-Bioconcentration factor (BCF); B3-Hydrolysis; B4-Atmospheric oxidation; B5-Biotransformation;
B6-Distribution between environmental compartments; B7-Bioaccumulation factor (BAF); B8-Half life in each medium; B9-Photodegradation;
B10-Biomagnification factor (BMF); C1-Aquatic toxicity; C2-Terrestrial toxicity; C3-Microbial inhibition; C4-Sediment toxicity; D1-Mutagenicity;
D2-Carcinogenicity; D3-Skin/respiratory sensitization; D4-Acute toxicity; D5-Skin irritation/corrosion; D6-Genotoxicity; D7-Eye damage/irritation;
D8-Developmental toxicity; D9-Toxicological signal pathways; D10-Hepatotoxicity; D11-Reproductive toxicity; D12-Cardiotoxicity;
D13-Repeated dose toxicity; D14-Neurotoxicity; D15-Immunotoxicity; D16-Photoinduced toxicity; E1-Metabolism; E2-DNA/protein binding;
E3-Blood-brain barrier penetration; E4-Skin penetration; E5-Gastrointestinal absorption; E6-Excretion.
化学品的部分参数值如Kow和大气氧化性等,与温度、pH值等环境条件相关。多数软件仅能预测单一环境条件下的参数值。在统计的软件中,ACD/Percepta考虑了pH值对可解离化学品Kow值的影响,可预测化学品在pH从1到9时的Kow值。CPTP软件可在用户设定的环境温度下,预测化学品与羟基自由基或臭氧的反应速率常数。
2.3 软件应用的方法
软件使用的预测方法、分子描述符和模型的应用域表征方法共同构成软件的方法体系,是影响其预测机理透明度和结果可靠性的重要因素。
2.3.1 预测方法
统计的计算毒理学软件中用到的预测手段可分为交叉参照(read-across, RA)、QSAR和专家系统3种(表2)。它们均基于“结构相似的化合物往往具有相似活性”的相似性原理,但应用于化学品管理时存在不同的优势和局限。
RA将化学品按相似性分组,再利用相似化合物的已知实验数据推测目标物对应的数据空缺,结果可靠性很大程度上取决于分组步骤的相似性判断依据[19]。统计中以RA方法为主的软件Toxmatch和OECD QSAR Toolbox都包含多种相似性度量方法。Toxmatch中包含了基于描述符的相似度指标,如Euclidean距离指数、Tanimoto和Hodgkin-Richards系数,同时包含了基于结构的相似度指标,如Tanimoto指数、Hellinger距离指数和最大公共子结构(maximum common substructure, MCS),以及多个针对毒性预测的预定义分类方案。OECD QSAR Toolbox软件支持用户根据预测终点选用不同特征定义相似物质并分组,包括了物质结构(特定结构碎片、活性基团、有机官能团或化学元素)、理化性质(性质参数如logKow、分子量的数值范围)、作用机制(警示结构)或毒理学效应(与生物大分子相互作用的大小)等[20-21]。RA软件不需要预先构建模型,但要求用户对与终点相关的机理有一定的认识,更适合具有专业背景的人使用。
QSAR模型基于特定的原始数据集(训练集),建立物质微观结构与其理化性质/环境行为/毒理效应参数的统计学关系,在化学品性质预测中得到了广泛的应用[22]。在统计的软件中,QSAR模型用到了多元线性回归、偏最小二乘回归、随机森林、逻辑回归、k近邻学习、支持向量机、神经网络等统计学方法和机器学习算法构建模型(表3)。训练集物质在结构或作用机制上的差异可能影响QSAR模型的预测性能。为了应对这一问题,Lazar软件将k近邻学习与支持向量机结合,在预测时自动筛选相似化合物作为训练集生成局部模型[23]。T.E.S.T.软件中也包含一种类似的局部多元线性回归模型[24]。UL’s Cheminformatics Tool Kit软件使用的RASAR模型,利用相似度作为特征向量构建随机森林模型,也增加了个体预测结果的置信度[25]。
专家系统是使用规则预测物质活性的方法,一般用到了“警示结构(structure alerts, SAs)”的概念。SAs指与物质毒性相关的化学结构[26]。统计的专家系统软件利用从文献中获取的专家知识(OncoLogic, Hazard Expert, Derek Nexus, Toxtree, Leadscope)或对原始数据集的统计分析(Case Ultra),构建阐明SAs与毒性关系的规则模型。基于引发活性的SAs建模,导致大部分专家系统难以识别非活性化合物,容易给出假阳性预测,因此在化学品风险评价中不能用于给出结论性的毒性预测,但可在化学品风险的分层评价策略中用于筛查毒性潜力,确定需优先进行in vitro测试的化合物[27]。除此之外,专家系统还可应用于RA,基于毒性作用机制进行物质分组;可与QSAR模型结合使用,进行模型的机理域表征,但这一功能在统计的软件中没有得到应用。
表2 软件的预测手段
Table 2 Prediction approaches of software

软件Software预测手段Prediction approachesQSARRA专家系统Expert systemOECD QSAR Toolbox√√√Danish QSAR Database/Models√VEGA√EPI Suite√ACD/Percepta√CASE Ultra√√ADMET Predictor√OPERA√T.E.S.T.√BIOVIA√ChemTunes/ToxGPS√√√Toxtree√Molcode Toolbox√Hazard Expert√Derek√Lazar√UL’s Cheminformatics Tool Kit√Leadscope Model Applier√√OncoLogic√ProTox Ⅱ√AdmetSAR 2√Toxmatch√√KATE√PBT Profiler√CPTP√
注:QSAR表示定量构效关系;RA表示交叉参照。
Note: QSAR is quantitative structure-activity relationship; RA is read-across.
2.3.2 分子描述符类型
分子描述符是关联分子结构与活性的重要媒介,是QSAR建模的核心。统计的软件普遍使用易于获取和计算的分子性质、组成、拓扑描述符或分子指纹表征分子特征(表4)。其中,分子性质为分子的理化参数,如正辛醇/水分配系数、水溶解度、分子量等,可通过实验或计算方式获取;组成描述符反映分子的化学成分,包括原子、键、环或结构片段的数量等;拓扑描述符基于表征分子图的矩阵计算获得,编码关于原子排列和连通性的信息[28-29];分子指纹是通过构造布尔或整型向量,描述子结构或碎片模式存在与否的分子结构表征方式,常用于化合物相似性搜索、聚类和分类[30]。相比之下,基于分子立体结构计算的描述符,如几何描述符、量化描述符,由于涉及复杂的构象优化过程,在软件中应用较少。其中,几何描述符可表征分子三维构象信息,能更深层次地阐释分子结构特征[28];量化描述符呈现分子的电子结构与能量信息,物理意义明确,可解释性强,适用于光解、大气降解、氧化还原等化学反应性主导的终点的预测[31],在未来有较大的应用潜力。
2.3.3 应用域表征方法
QSAR和专家系统模型都仅在一定的化学空间内适用,该空间被称为应用域[22]。部分软件能给出预测是否在应用域内的提示,以帮助用户评估预测结果的可靠性。
统计中的部分软件采用基于描述符空间范围(描述符域)或/和目标物与训练集化合物的结构相似性(结构域)的方法表征模型应用域[32]。其中,软件采用的描述符域表征方法普遍为考察描述符取值范围,OPERA和CPTP软件采用了基于杠杆值计算目标物与训练集在描述符空间中的距离的方法[33-34]。软件的结构域表征方法主要为计算结构相似性指数,如Tanimoto系数。EPI Suite、BIOVIA和VEGA采用了筛查比对训练集与目标物结构特征的方法,当目标物中出现训练集未包括的子结构时,判断为在模型结构域外(表5)。对于宏观毒性效应,分子结构细微的变化也可能引起活性的突变[28],致使前2种应用域表征方法无法保证预测的可靠性。ACD/Percepta、T.E.S.T.和VEGA可通过考察模型对相似物的预测性能识别这种情况,隐含了对模型机理域的考虑[35]。
表3 软件中QSAR模型使用的建模方法
Table 3 Modeling techniques used by QSAR models in software
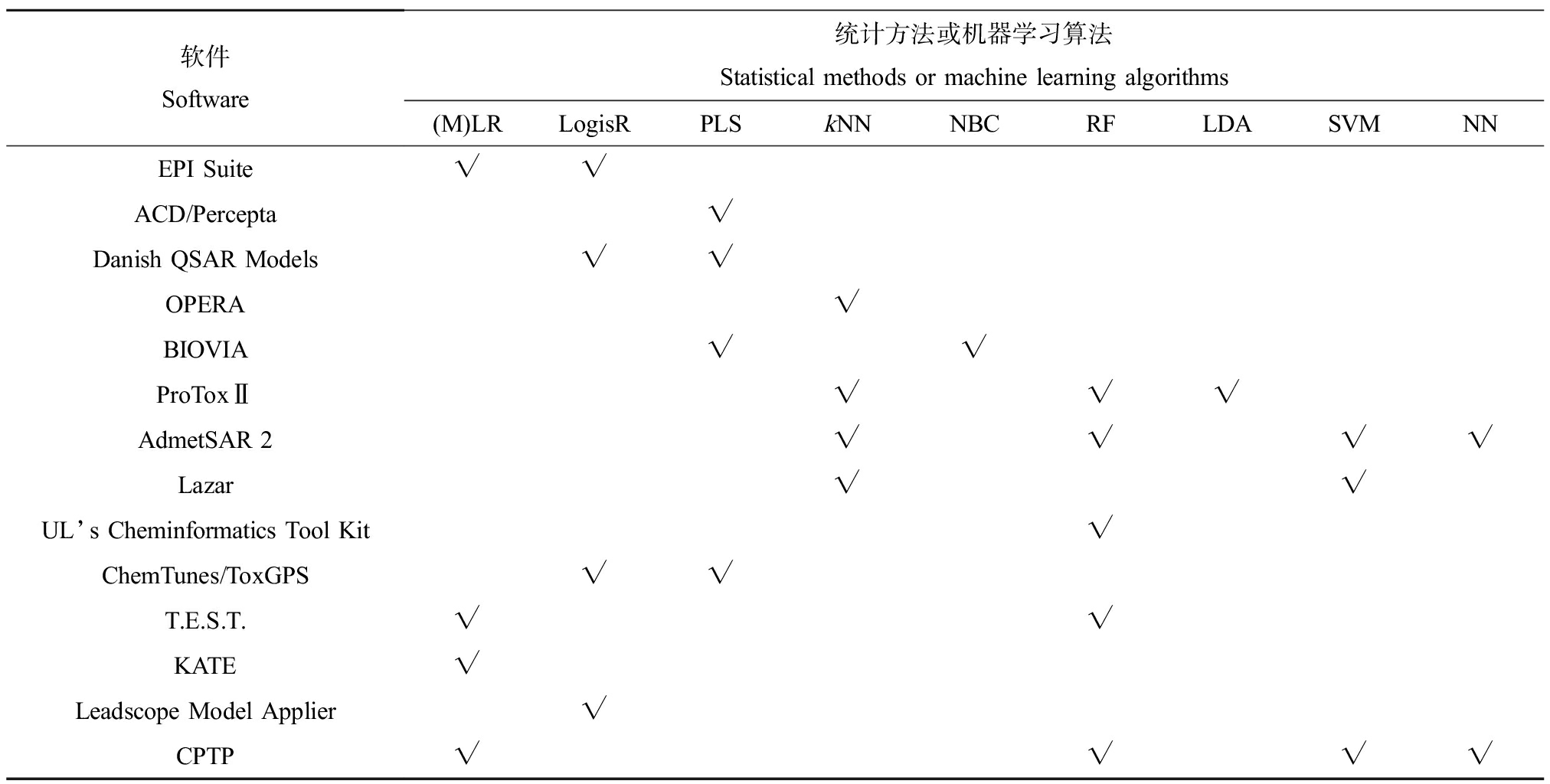
软件Software统计方法或机器学习算法Statistical methods or machine learning algorithms(M)LRLogisRPLSkNNNBCRFLDASVMNNEPI Suite√√ACD/Percepta√Danish QSAR Models√√OPERA√BIOVIA√√ProToxⅡ√√√AdmetSAR 2√√√√Lazar√√UL’s Cheminformatics Tool Kit√ChemTunes/ToxGPS√√T.E.S.T.√√KATE√Leadscope Model Applier√CPTP√√√√
注:(M)LR表示(多元)线性回归;LogisR表示逻辑回归;PLS表示偏最小二乘法;kNN表示k近邻学习方法;NBC表示朴素贝叶斯分类;RF表示随机森林;LDA表示线性判别分析;SVM表示支持向量机;NN表示神经网络。
Note: (M)LR is (Multiple) Liner Regression; LogisR is Logistic Regression; PLS is Partial Least Squares; kNN is k-Nearest Neighbor; NBC is Naive Bayesian Classifier; RF is Random Forest; LDA is Linear Discriminative Analysis; SVM is Support Vector Machine; NN is Neural Network.
早期的专家系统软件大多以引发SAs作为预测在应用域内的判断依据。但警示结构也只有在特定的化学结构空间内才有效,导致模型常给出假阳性的预测。Derek、OECD QSAR Toolbox和Case Ultra软件中通过加入判断警示结构应用域的方法,增强了模型的预测性能[36-38]。
2.4 软件实际操作
2.4.1 软件功能
统计的软件都支持多种输入形式,具有将CAS号或分子平面图转换为简化分子线性输入规范(simplified molecular input line entry system, SMILES)的功能。部分软件允许以不同形式的文件下载预测结果。其中,OECD QSAR Toolbox能生成符合欧洲化学品管理局(ECHA)官方要求的结论报告,直接用于物质申报。部分软件能实现化合物的批量处理和多终点预测,提高了软件使用效率(表6)。
除了基本的预测功能外,部分软件还包含了特殊功能,进一步提升软件结果科学性、用户友好度、或满足用户多方面的需求。Danish QSAR Database/Models和AdmetSAR 2提供了数据库查询功能,AdmetSAR 2还能辅助用户进行分子优化,拓展了其在化学品研发中的应用。基于警示结构的专家系统软件Toxtree支持用户对软件中的决策树进行查询和修改,并拥有自定义决策树的功能。利用RA进行预测的OECD QSAR Toolbox软件,由物质识别、物质分析、物质分类、数据收集、预测和报告六大模块构成了一套可供用户自主操作的工作流程,允许用户自选数据库、相似性判断依据和数据填充方法,还支持人为替换趋势分析公式中的自变量,为具有专业背景的用户提供更好的服务。
表4 软件使用的分子描述符
Table 4 Molecular descriptors used in the software

软件Software分子描述符类型Types of molecular descriptors分子性质描述符Molecular properties descriptors组成描述符Constitutional descriptors拓扑描述符Topologic descriptors几何描述符Geometric descriptors量子化学描述符Quantum chemistry descriptors分子指纹Molecular fingerprintsEPI Suite√√PBT Profiler√√ACD/Percepta√√Danish QSAR Models√√OPERA√√BIOVIA√√√ProToxⅡ√√AdmetSAR 2√√√Lazar√√√ChemTunes/ToxGPS√√√√√Toxmatch√√√√√T.E.S.T.√√√VEGA√√√KATE√UL’s Cheminformatics Tool Kit√OECD QSAR Toolbox√√Leadscope Model Applier√√CASE Ultra√√Molcode Toolbox√CPTP√√√√
表5 应用域表征方法
Table 5 Methods used to characterize the applicability domain

软件Software应用域表征方法Applicability domain characterization methods描述符范围Descriptor ranges描述符空间距离指标Descriptor space distance matrix结构相似性度量指标Structure similarity matrix结构特征筛查Structure feature screening类似物预测性能Analogue prediction performanceEPI Suite√√ACD/Percepta√Danish QSAR Models√OPERA√√BIOVIA√√ProToxⅡ√AdmetSAR 2√Lazar√UL’s Cheminformatics Tool Kit√T.E.S.T.√VEGA√√√√KATE√CPTP√√
表6 软件功能比较
Table 6 Comparison of software functions

软件Software输入形式Input forms结果下载形式Result download format批量预测Batch predictions多终点同时预测Simultaneous predictions of multiple endpointsEPI SuiteCAS,SMILESPDF√√OECD QSAR ToolboxCAS,SMILESPDFT.E.S.T.CAS,分子绘图CAS, molecule drawingN/A√ToxtreeSMILES,分子绘图SMILES, molecule drawingN/A√LazarSMILES,分子绘图SMILES, molecule drawingN/A√OncoLogicCAS,化学名称CAS, chemical nameRTFVEGASMILESPDF, CSV√√Danish QSAR ModelsSMILES,分子绘图,化学名称,MOL/SDF文件 SMILES, molecule drawing, chemical name, MOL/SDF filePDF√OPERASMI/MOL/SDF/TXT文件SMI/MOL/SDF/TXT fileCSV, TXT√ProToxⅡSMILES,分子绘图,化学名称SMILES, molecule drawing, chemical nameN/A√AdmetSAR 2SMILES,分子绘图SMILES, molecule drawingCSV√√CPTPCAS,SMILES,化学名称,分子绘图CAS, SMILES, chemical name, molecule drawingN/A√√
2.4.2 软件的信息完整度
面向化学品风险评价的计算毒理学软件作为管理中的决策支持工具,有必要对使用的模型和预测结果不确定性提供详细的信息,以帮助用户对模型有效性和预测结果可靠性进行评估。12款可免费获取的软件提供信息的情况如表7所示。
研究以OECD的QSAR验证导则为依据,考察各软件的模型信息完整度。根据导则对用于管理活动的QSAR模型的要求,软件应对其使用的QSAR模型同时提供明确定义的预测终点、明确的算法、模型应用域、模型性能的度量以及尽可能提供模型的机理解释[14]。另外,考虑到建模数据质量和数量对模型性能的影响[39],是否提供建模原始数据也被纳入考察范围。统计中,EPI Suite、VEGA、OECD QSAR Toolbox包含模型信息较为完整,但部分软件存在信息的缺失,可能限制其在管理中的实际应用。软件可通过给出应用域评估、目标物实验值及类似物数据,帮助用户评估预测结果的不确定性。统计中,OECD QSAR Toolbox、T.E.S.T.、Lazar、VEGA和OPERA同时给出了这3种信息,EPI Suite、Toxtree、OncoLogic、AdmetSAR 2和CPTP软件给出相关信息较少。此外,部分软件通过提供开发背景信息发挥软件的科普功能,通过提供用户手册或界面操作提示等方式扫除用户的使用障碍,增强了软件对非专业用户的友好度。
2.4.3 QSAR软件预测准确度比较
表8统计了7款软件中具有代表性的QSAR模型的预测性能指标。在回归模型中,各软件的正辛醇/水分配系数和生物富集因子模型对验证集和测试集的R2均在0.8左右,具有较好的预测能力,然而鱼类急性毒性模型的R2均在0.7以下,预测性能相对较低。各软件中生物降解性和致突变性2种分类模型的准确度均在0.8左右,具有较好的分类效果。
将软件对“百菌清”3个终点(endpoint)的预测结果与实验值比较(表9),各软件对LC50的预测结果差异大且均与文献值有较大差距,表明上述软件对水生生物毒性的预测性能有待提高。正辛醇水分配系数(logKow)和生物富集因子(logBCF)的预测值与实验值接近,其中CPTP对logKow的预测值和EPI Suite、VEGA对logBCF的预测与实验值最接近。
2.5 软件综合推荐
综合用户友好度、终点覆盖数量、信息全面度和软件功能对实际操作的12款软件进行评价,推荐目前在化学品管理中可使用EPI Suite、VEGA和OECD QSAR Toolbox软件。这3款软件都具有操作简单、界面友好、覆盖终点数量较多、模型信息全面的特点。其中EPI Suite和VEGA软件可实现批量预测和多终点同时预测,适合辅助监管机构开展大量化学品的风险评价;OECD QSAR Toolbox具有极高操作自由度,可提供详细的预测报告,适合辅助企业的化学品注册登记时所需的信息申报。
3 讨论(Discussion)
3.1 计算毒理学软件的未来进步空间
根据统计分析,所搜集的软件在预测终点覆盖度、预测结果可靠性、应用方法先进性和实用性等方面仍存在局限。软件对部分值得关注的终点,如环境光化学降解性、陆生生物积累性的覆盖较少,对不同终点的预测性能存在差异,且大多未能考虑环境条件对性质参数的影响。统计软件中对三维分子描述符及复杂模型算法,如深度神经网络的应用较少,未能发挥新兴建模技术在大数据分析和预测性能提升方面的应用潜力[40-41]。未来应逐渐扩充软件的预测终点,积累高质量的数据资源作为建模原始数据,扩增软件对三维描述符的运算和存储功能,并将深度神经网络等先进方法应用于软件模型的构建,以提升软件的预测性能,并扩大其应用范围。此外,还应保证软件的用户友好度和信息完整度,以引导公众探索兴趣,增强专业用户使用信心,以及支持化学品管理决策。
在功能方面,软件可支持用户发展自定义的预测模型,还可提供数据查询等拓展功能,以满足用户多方面的需求。可将多介质环境模型、毒代动力学模型和系统生物学模型等纳入软件的模型体系,结合软件的参数预测功能,实现对化学品从暴露到导致危害效应连续过程的模拟预测。另外,除了作为性质预测工具,计算毒理学软件还可纳入集成评估框架和决策分析工具,形成集预测、评估和决策分析于一体的智能决策支持系统,在未来的化学品管理活动中发挥更大的作用。
表7 软件信息完整度比较
Table 7 Comparison of software information comprehensiveness
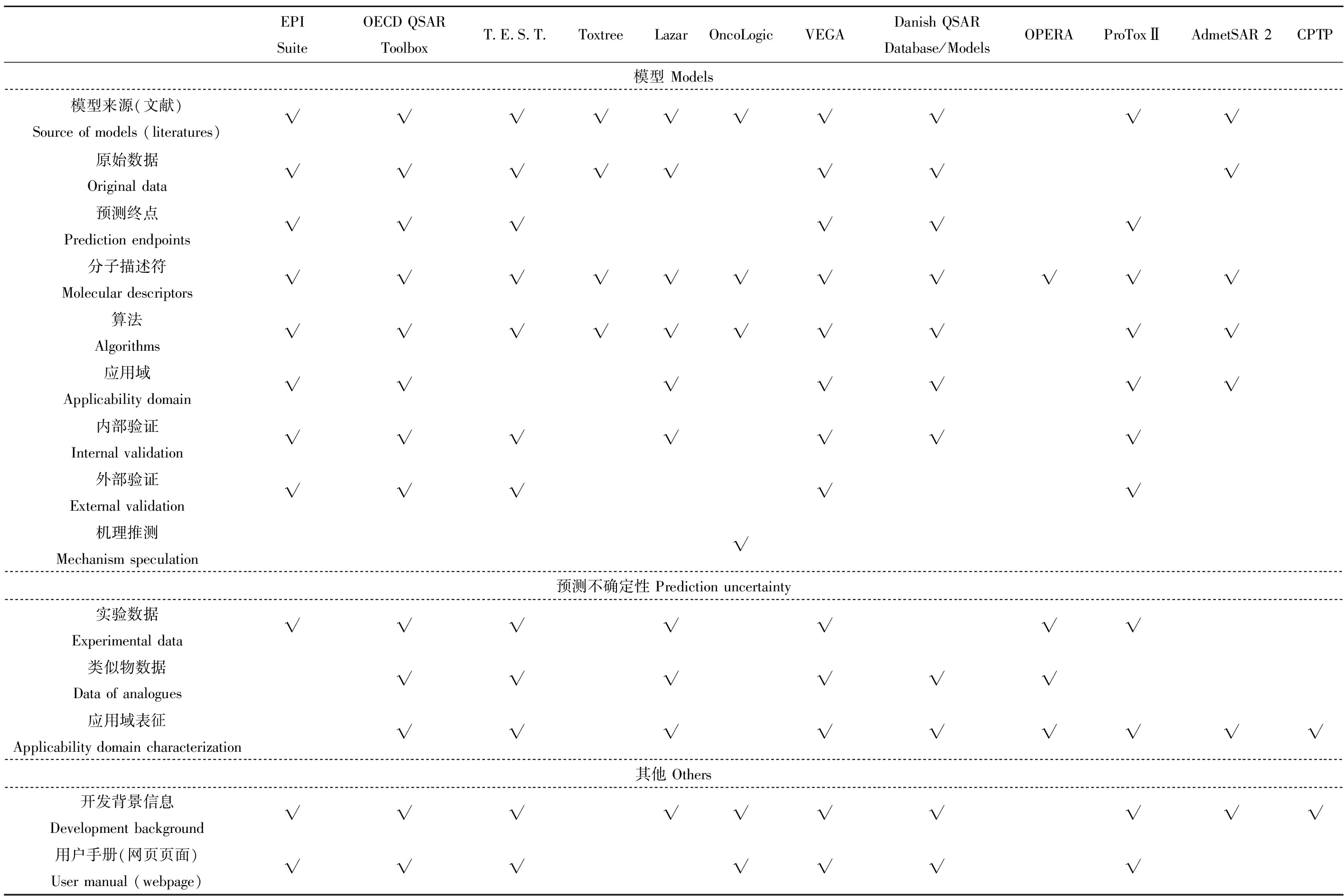
EPI SuiteOECD QSAR ToolboxT.E.S.T.ToxtreeLazarOncoLogicVEGADanish QSAR Database/ModelsOPERAProToxⅡAdmetSAR 2CPTP模型 Models模型来源(文献)Source of models (literatures)√√√√√√√√√√原始数据Original data√√√√√√√√预测终点Prediction endpoints√√√√√√分子描述符Molecular descriptors√√√√√√√√√√√算法Algorithms√√√√√√√√√√应用域Applicability domain√√√√√√√内部验证Internal validation√√√√√√√外部验证External validation√√√√√机理推测Mechanism speculation√预测不确定性 Prediction uncertainty实验数据Experimental data√√√√√√√类似物数据Data of analogues√√√√√√应用域表征Applicability domain characterization√√√√√√√√√其他 Others开发背景信息Development background√√√√√√√√√√用户手册(网页页面)User manual (webpage)√√√√√√√
表8 软件中代表性QSAR模型的预测性能
Table 8 Predictive performance of representative QSAR models in the software

软件SoftwareQSAR模型的验证指标QSAR model validation index正辛醇/水分配系数Octanol-water partition coefficient鱼类急性毒性Fish acute toxicity生物富集因子Bioconcentration factor生物降解性Biodegradation致突变性MutagenicityEPI SuiteTr: R2=0.98Te: R2=0.94N/ATr: R2=0.83Te: R2=0.82Tr: A=0.82Te: A=0.81N/AVEGATe: R2=0.84, RMSE=0.72Tr: R2=0.69, RMSE=0.83Te: R2=0.64, RMSE=0.89Tr: R2=0.8, RMSE=0.55Te: R2=0.79, RMSE=0.66Tr: A=0.92, Sen=0.90, Spe=0.95Te: A=0.82, Sen=0.77, Spe=0.87Tr: A=0.92, Sen=0.97, Spe=0.86Te: A=0.83, Sen=0.90, Spe=0.74T.E.S.T.N/ATe: R2=0.71, RMSE=0.80, MAE=0.57Te: R2=0.76, RMSE=0.66, MAE=0.51N/ATe: A=0.79, Sen=0.79, Spe=0.79LazarN/ATr: R2=0.51, RMSE=0.92, MAE=0.68N/AN/ATr: A=0.82, Sen=0.86, Spe=0.85Danish QSAR DB与EPI Suite使用同一模型Use the same model as EPI Suite未提供信息No information provided与EPI Suite使用同一模型Use the same model as EPI SuiteTr: A=0.86, Sen=0.87, Spe=0.85Tr: A=0.85, Sen=0.84, Spe=0.86Te: A=0.82, Sen=0.78, Spe=0.84ProToxⅡN/AN/AN/AN/ATe: R2=0.84OPERATr: R2=0.86, RMSE=0.67Te: R2=0.86, RMSE=0.79N/ATr: R2=0.84, RMSE=0.55Te: R2=0.83, RMSE=0.64N/AN/A
注:Tr表示训练集;Te表示验证集; R2表示决定系数;RMSE表示均方根误差;MAE表示平均绝对误差;A表示准确度;Sen表示灵敏度;Spe表示特异性;N/A表示软件没有相应的模型。
Note: Tr is training set; Te is test set; R2 is the coefficient of determination; RMSE is the root mean squared error; MAE is the mean absolute error; A means the prediction accuracy; Sen means the prediction sensitivity; Spe means the prediction specificity; N/A denotes that the software has no corresponding models.
表9 百菌清代表性终点的实验值和预测值比较
Table 9 Comparison of experimental and predicted values of the representative endpoints of chlorothalonil
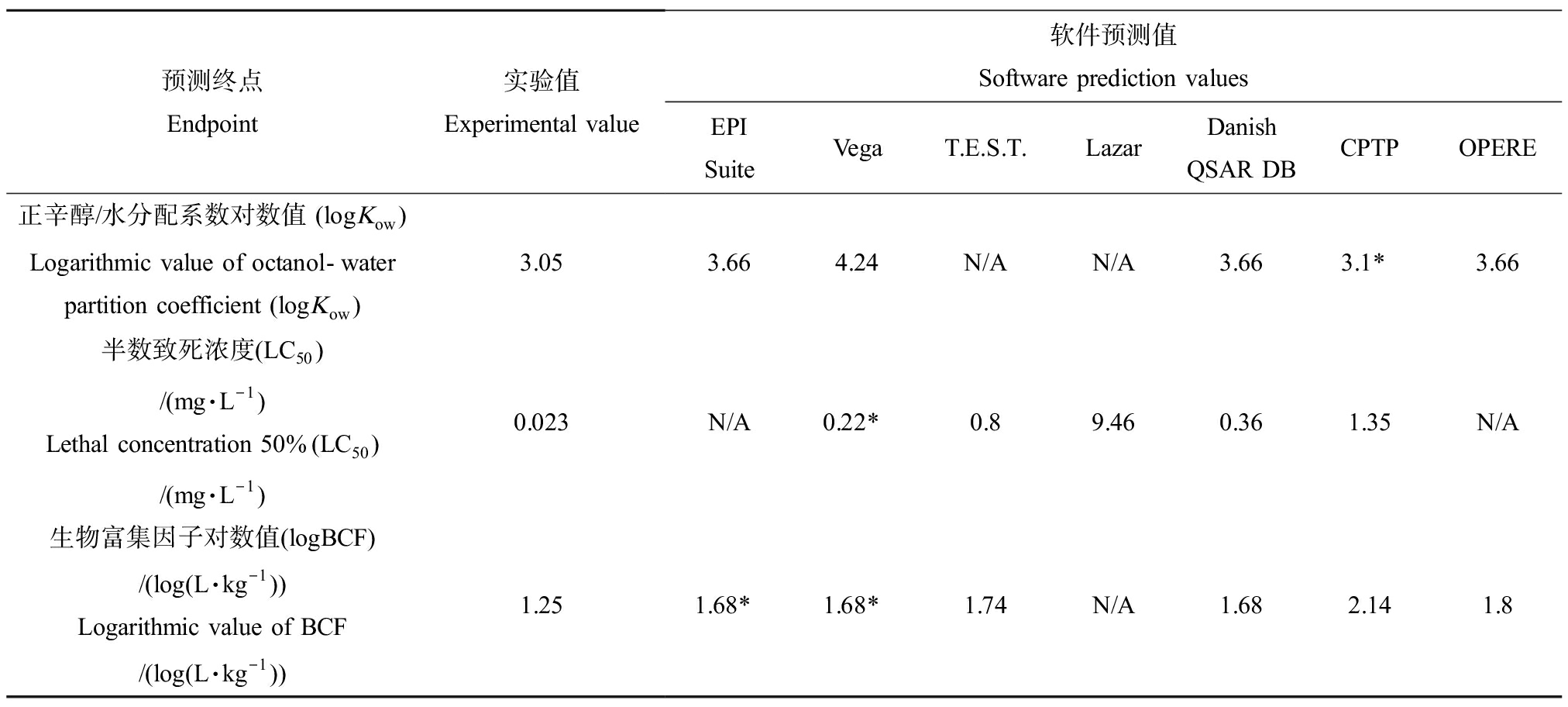
预测终点Endpoint实验值Experimental value软件预测值Software prediction valuesEPI SuiteVegaT.E.S.T.LazarDanish QSAR DBCPTPOPERE正辛醇/水分配系数对数值 (logKow)Logarithmic value of octanol-water partition coefficient (logKow)3.053.664.24N/AN/A3.663.1*3.66半数致死浓度(LC50)/(mg·L-1)Lethal concentration 50%(LC50)/(mg·L -1)0.023N/A0.22*0.89.460.361.35N/A生物富集因子对数值(logBCF) /(log(L·kg -1))Logarithmic value of BCF/(log(L·kg-1))1.251.68*1.68*1.74N/A1.682.141.8
注:N/A表示软件没有相应模型;*表示与实验值最接近的预测值;Kow为正辛醇/水分配系数;LC50为半数致死浓度;BCF为生物富集因子。
Note: N/A represents the software has no corresponding models; *represents the predicted value closest to the experimental value; Kow is the octanol-water partition coefficient; LC50 is the lethal concentration 50%; BCF is the bioconcentration factor.
3.2 我国的化学品风险预测软件开发
欧美国家较早开始重视计算毒理学工具在化学品风险管理中的应用,在法规层面上将其纳入化学品风险评价的方法体系,并通过社会各界合作交流促进了信息共享和多学科的融合,为计算毒理学软件的开发创造了良好的条件。这些软件又为欧美国家的化学品管理提供了信息化技术支持。与国外相比,我国的计算毒理学研究还处于起步阶段,相关技术的应用潜力未能得到充分发挥。近年来,我国的化学品生产规模不断扩大,化学品管理任务日益艰巨,因此急需借鉴美国和欧盟的成功经验,推进计算毒理学研究和预测工具的开发和应用,以辅助化学品风险评价和管理工作的开展。结合我国的实际情况,提出以下几点建议。
(1)健全我国化学品管理法规,形成对新化学物质和市场现有化学品的全面风险管理,增加化学品管理中应用计算毒理学技术的需求。
(2)制定化学品风险预测技术方法规范和导则,指导实用性预测工具的开发与应用。
(3)加强交叉学科人才培养及多学科合作交流平台的搭建,推动计算毒理学研究团队的组建与发展。
(4)加强学术界与政府、企业的合作。政府通过政策和资金方面的支持,鼓励高校及科研机构面向国家化学品管理与相关企业进行化学品注册登记、绿色设计等需求,有针对性、系统性地进行计算毒理学研究及相关实用工具的研发。
(5)加强社会各界及化学品管理机构之间的信息共享,探索数据持有者与模型开发者的数据共享的机制,鼓励研究机构搭建大型的模型库或模型网络共享平台,为软件开发积累数据和模型资源。
通讯作者简介:傅志强(1989—),男,博士,主要研究方向为环境新污染物的代谢转化行为及毒理效应的模拟预测。
[1] United Nations Environment Programme (UNEP). Global Chemicals Outlook Ⅱ: From legacies to innovative solutions: Implementing the 2030 agenda for sustainable development [R]. Geneva: UNEP, 2019
[2] Landrigan P J, Fuller R, Acosta N J R, et al. The Lancet Commission on pollution and health [J]. Lancet, 2018, 391(10119): 462-512
[3] Wu S, Powers S, Zhu W, et al. Substantial contribution of extrinsic risk factors to cancer development [J]. Nature, 2016, 529(7584): 43-47
[4] 王中钰, 陈景文, 乔显亮, 等. 面向化学品风险评价的计算(预测)毒理学[J]. 中国科学: 化学, 2016, 46(2): 222-240
Wang Z Y, Chen J W, Qiao X L, et al. Computational toxicology: Oriented for chemicals risk assessment [J]. Scientia Sinica Chimica, 2016, 46(2): 222-240 (in Chinese)
[5] Wang Z Y, Walker G W, Muir D C G, et al. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories [J]. Environmental Science & Technology, 2020, 54(5): 2575-2584
[6] Schmidt C W. TSCA 2.0: A new era in chemical risk management [J]. Environmental Health Perspectives, 2016, 124(10): A182-A186
[7] Krimsky S. The unsteady state and inertia of chemical regulation under the US Toxic Substances Control Act [J]. PLoS Biology, 2017, 15(12): e2002404
[8] Hartung T, Rovida C. Chemical regulators have overreached [J]. Nature, 2009, 460(7259): 1080-1081
[9] Kavlock R, Dix D. Computational toxicology as implemented by the US EPA: Providing high throughput decision support tools for screening and assessing chemical exposure, hazard and risk [J]. Journal of Toxicology and Environmental Health, Part B, 2010, 13(2-4): 197-217
[10] Egeghy P P, Sheldon L S, Isaacs K K, et al. Computational exposure science: An emerging discipline to support 21st-Century risk assessment [J]. Environmental Health Perspectives, 2016, 124(6): 697-702
[11] Collins F S, Gray G M, Bucher J R. Toxicology. Transforming environmental health protection [J]. Science, 2008, 319(5865): 906-907
[12] Pavan M, Worth A P. Publicly-accessible QSAR software tools developed by the Joint Research Centre [J]. SAR and QSAR in Environmental Research, 2008, 19(7-8): 785-799
[13] Jaworska J S, Comber M, Auer C, et al. Summary of a workshop on regulatory acceptance of (Q)SARs for human health and environmental endpoints [J]. Environmental Health Perspectives, 2003, 111(10): 1358-1360
[14] Organization for Economic Co-Operation and Development (OECD). Guidance document on the validation of (quantitative) structure-activity relationships [(Q)SARs] models [R]. Paris: OECD, 2007
[15] European Commission, Joint Research Centre (JRC). JRC QSAR Model Database [DB]. Brussels: European Commission, 2020
[16] Blaauboer B J. Biokinetic modeling and in vitro-in vivo extrapolations [J]. Journal of Toxicology and Environmental Health, Part B, 2010, 13(2-4): 242-252
[17] MacKay D. Finding fugacity feasible [J]. Environmental Science & Technology, 1979, 13(10): 1218-1223
[18] LaLone C A, Ankley G T, Belanger S E, et al. Advancing the adverse outcome pathway framework: An international horizon scanning approach [J]. Environmental Toxicology and Chemistry, 2017, 36(6): 1411-1421
[19] Ball N, Cronin M T, Shen J, et al. Toward good read-across practice (GRAP) guidance [J]. ALTEX, 2016, 33(2): 149-166
[20] Patlewicz G, Jeliazkova N, Gallegos Saliner A, et al. Toxmatch: A new software tool to aid in the development and evaluation of chemically similar groups [J]. SAR and QSAR in Environmental Research, 2008, 19(3-4): 397-412
[21] Dimitrov S D, Diderich R, Sobanski T, et al. QSAR toolbox: Workflow and major functionalities [J]. SAR and QSAR in Environmental Research, 2016, 27(3): 203-219
[22] 陈景文, 李雪花, 于海瀛, 等. 面向毒害有机物生态风险评价的(Q)SAR技术: 进展与展望[J]. 中国科学(B辑: 化学), 2008, 38(6): 461-474
[23] Maunz A, Gütlein M, Rautenberg M, et al. Lazar: A modular predictive toxicology framework [J]. Frontiers in Pharmacology, 2013, 4: 38
[24] Contrera J F, Matthews E J, Benz R D. Predicting the carcinogenic potential of pharmaceuticals in rodents using molecular structural similarity and E-state indices [J]. Regulatory Toxicology and Pharmacology, 2003, 38(3): 243-259
[25] Luechtefeld T, Marsh D, Rowlands C, et al. Machine learning of toxicological big data enables read-across structure activity relationships (RASAR) outperforming animal test reproducibility [J]. Toxicological Sciences: An Official Journal of the Society of Toxicology, 2018, 165(1): 198-212
[26] Raies A B, Bajic V B. In silico toxicology: Computational methods for the prediction of chemical toxicity [J]. Wiley Interdisciplinary Reviews: Computational Molecular Science, 2016, 6(2): 147-172
[27] European Centre for Ecotoxicology and Toxicology of Chemicals (ECETOC). (Q)SARs: Evaluation of the commercially available software for human health and environmental endpoints with respect to chemical management applications [R]. Brussels: ECETOC, 2003
[28] Lo Y C, Rensi S E, Torng W, et al. Machine learning in chemoinformatics and drug discovery [J]. Drug Discovery Today, 2018, 23(8): 1538-1546
[29] Vo A H, van Vleet T R, Gupta R R, et al. An overview of machine learning and big data for drug toxicity evaluation [J]. Chemical Research in Toxicology, 2020, 33(1): 20-37
[30] Rogers D, Hahn M. Extended-connectivity fingerprints [J]. Journal of Chemical Information and Modeling, 2010, 50(5): 742-754
[31] Mamy L, Patureau D, Barriuso E, et al. Prediction of the fate of organic compounds in the environment from their molecular properties: A review [J]. Critical Reviews in Environmental Science and Technology, 2015, 45(12): 1277-1377
[32] Netzeva T I, Worth A, Aldenberg T, et al. Current status of methods for defining the applicability domain of (quantitative) structure-activity relationships. The report and recommendations of ECVAM Workshop 52 [J]. Alternatives to Laboratory Animals, 2005, 33(2): 155-173
[33] Mansouri K, Grulke C M, Judson R S, et al. OPERA models for predicting physicochemical properties and environmental fate endpoints [J]. Journal of Cheminformatics, 2018, 10(1): 10
[34] Todeschini R, Ballabio D, Consonni V, et al. Locally centred Mahalanobis distance: A new distance measure with salient features towards outlier detection [J]. Analytica Chimica Acta, 2013, 787: 1-9
[35] Maggiora G M. On outliers and activity cliffs: Why QSAR often disappoints [J]. Journal of Chemical Information and Modeling, 2006, 46(4): 1535
[36] Williams R V, Amberg A, Brigo A, et al. It’s difficult, but important, to make negative predictions [J]. Regulatory Toxicology and Pharmacology, 2016, 76: 79-86
[37] Chakravarti S K, Saiakhov R D, Klopman G. Optimizing predictive performance of CASE Ultra expert system models using the applicability domains of individual toxicity alerts [J]. Journal of Chemical Information and Modeling, 2012, 52(10): 2609-2618
[38] Yordanova D, Schultz T W, Kuseva C, et al. Alert performance: A new functionality in the OECD QSAR Toolbox [J]. Computational Toxicology, 2019, 10: 26-37
[39] Tropsha A. Best practices for QSAR model development, validation, and exploitation [J]. Molecular Informatics, 2010, 29(6-7): 476-488
[40] Min S, Lee B, Yoon S. Deep learning in bioinformatics [J]. Briefings in Bioinformatics, 2017, 18(5): 851-869
[41] Linden L, Goss K U, Endo S. 3D-QSAR predictions for bovine serum albumin-water partition coefficients of organic anions using quantum mechanically based descriptors [J]. Environmental Science Processes & Impacts, 2017, 19(3): 261-269